每年,全球约60万人死于疟疾,其中多数是非洲的五岁以下儿童。这种传染病由恶性疟原虫引起,会引发脑疟疾等致命并发症。而生活在疟疾流行区的人会逐渐产生免疫力,血液中存在能阻断疟疾的保护性抗体。这些人体内天然产生的保护性抗体像是一个宝藏,如果有技术能把这些抗体测出来,就可以将其用于疫苗或药物研发,相当于掌握了打开人体天然抗体这个宝藏大门的钥匙。
2025年8月20日,一项刚刚发表在PNAS的研究就验证了这个思路:快序生物的加拿大公司Rapid Novor和来自丹麦哥本哈根大学、美国斯克利普斯研究所、坦桑尼亚国家医学研究所组成的跨国研究团队,通过快序自主研发的多抗测序技术,直接从一名疟疾暴露儿童的血清多抗中,“钓”出了一组能广泛抑制疟原虫致病蛋白的抗体[1]。这一突破充分验证了快序直接从人血多抗蛋白中解析天然功能抗体序列的技术实力。快序这项技术的应用前景,绝不仅限于传染病防治,也为将来的自免和癌症药物的研发提供了全新的路径。
更令人振奋的是,这是继Rapid Novor快序生物于2024年10月在Nature Communications发表从新冠疫苗接种者的血清多抗中发现中和抗体的研究成果后[2],其血清多抗测序技术在顶级学术期刊发布的又一成功案例。本研究将人体多抗从头测序的应用场景,从疫苗接种者血清直接拓展至真实世界的康复患者血清——后者的发病和采血时间不可控,功能性抗体的浓度也更低,却具有不可替代的临床与转化价值。这一成果标志着快序通过血清多抗蛋白测序进行抗体发现路径已经成熟,也进一步彰显了快序生物在蛋白从头测序技术领域的绝对领跑地位。
疟疾的致病机制:PfEMP1蛋白介导的黏附
要理解这项研究的意义,得先从疟疾的致病机制说起。当恶性疟原虫侵入人体红细胞后,会在细胞表面表达一种名为PfEMP1(恶性疟原虫红细胞膜蛋白1)的蛋白。这种蛋白能牢牢抓住血管内皮细胞上的受体,如EPCR(内皮蛋白C受体),让红细胞黏附在血管里不被脾脏清除。大量被感染的红细胞堵塞毛细血管,会导致脑、肺等重要器官血流受阻,引发炎症和组织损伤,最终发展为脑型疟疾等严重病症,死亡率极高。
研究发现,PfEMP1家族存在许多变体,其中含CIDRα1结构域的变体最危险,它们能精准结合EPCR受体,是导致重症疟疾的“主力”。幸运的是,人体免疫系统会针对CIDRα1产生抗体,这些抗体能切断PfEMP1与EPCR的连接,阻止红细胞黏附。
但问题来了:CIDRα1家族成员众多,序列差异极大,因此很难找到能“通杀”多种变体的广谱抗体。更麻烦的是,传统方法从人体内寻找这些抗体,需要分离血液中的B细胞,通过噬菌体筛选或单B细胞测序等方法寻找有效抗体,而血液中分泌抗体的浆细胞极少,因此筛选难度极大,流程繁琐,耗时较长且成功率没有保障。
从血清多抗蛋白中直接解析抗体序列
研究团队从坦桑尼亚疟疾流行区的疟疾暴露者中,筛选出了血清中CIDRα1抗体反应最强的个体,通过抗原亲和纯化,获得了对CIDRα1变体具有广泛抑制性,且能阻断与EPCR结合的IgG多抗。接下来,通过多抗测序技术对血清抗体蛋白进行测序,成功解析了天然血清抗体中的单抗序列(图1)。
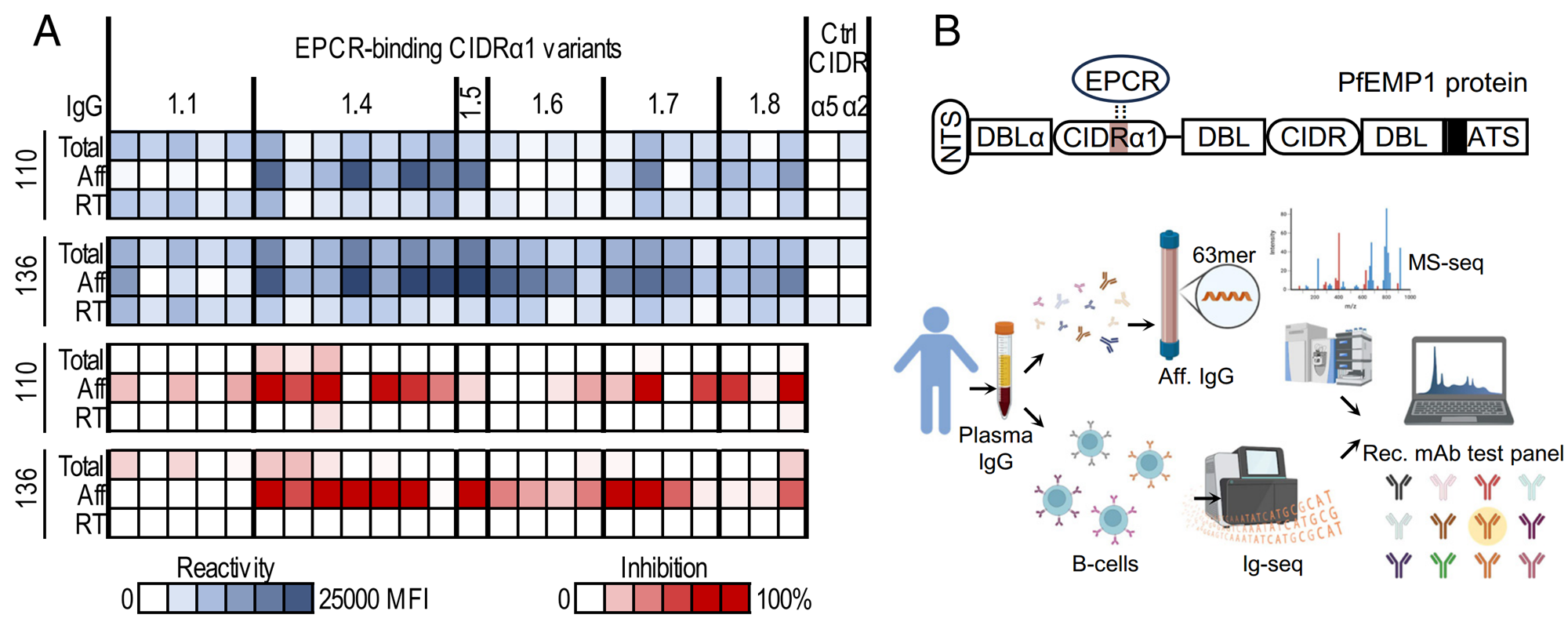
图 1. 多抗测序结合转录组分析血清抗体
结合CIDRα1的广谱中和抗体筛选
从110号样本亲和纯化富集的IgG中发现了3种抗体,其中存在一个克隆家族110-3(IGHV1-69基因起源,包含8个IGHV变体,CDR3区域序列高度保守),提示它们可能为同一B细胞克隆的体细胞突变产物。将这8个重链变体与相同110-3轻链配对重组表达后,研究人员发现它们均广泛结合CIDRα1.4-8(13/19),且对EPCR结合有强抑制作用(>50%)。进一步对110-3.4和110-3.6抗体(CDR3存在一个丙氨酸/脯氨酸差异)的分析显示:二者的生物膜干涉(BLI)结果表明他们对CIDRα1变体具有高亲和力(解离速率极慢);Luminex中和能力测定结果显示其对13种CIDRα1与EPCR结合的平均半数抑制浓度(IC50)分别为0.79 μg/mL和0.34 μg/mL,与此前发现的广谱抗体C7(0.1 μg/mL)、C74(0.38 μg/mL)相当。在疟原虫感染红细胞模型中,110-3.4能有效抑制表达CIDRα1.4(HB3VAR03)和CIDRα1.6(IT4VAR18)的红细胞与EPCR的黏附,效果与阳性对照(重组可溶性 EPCR)相当(图 2)。
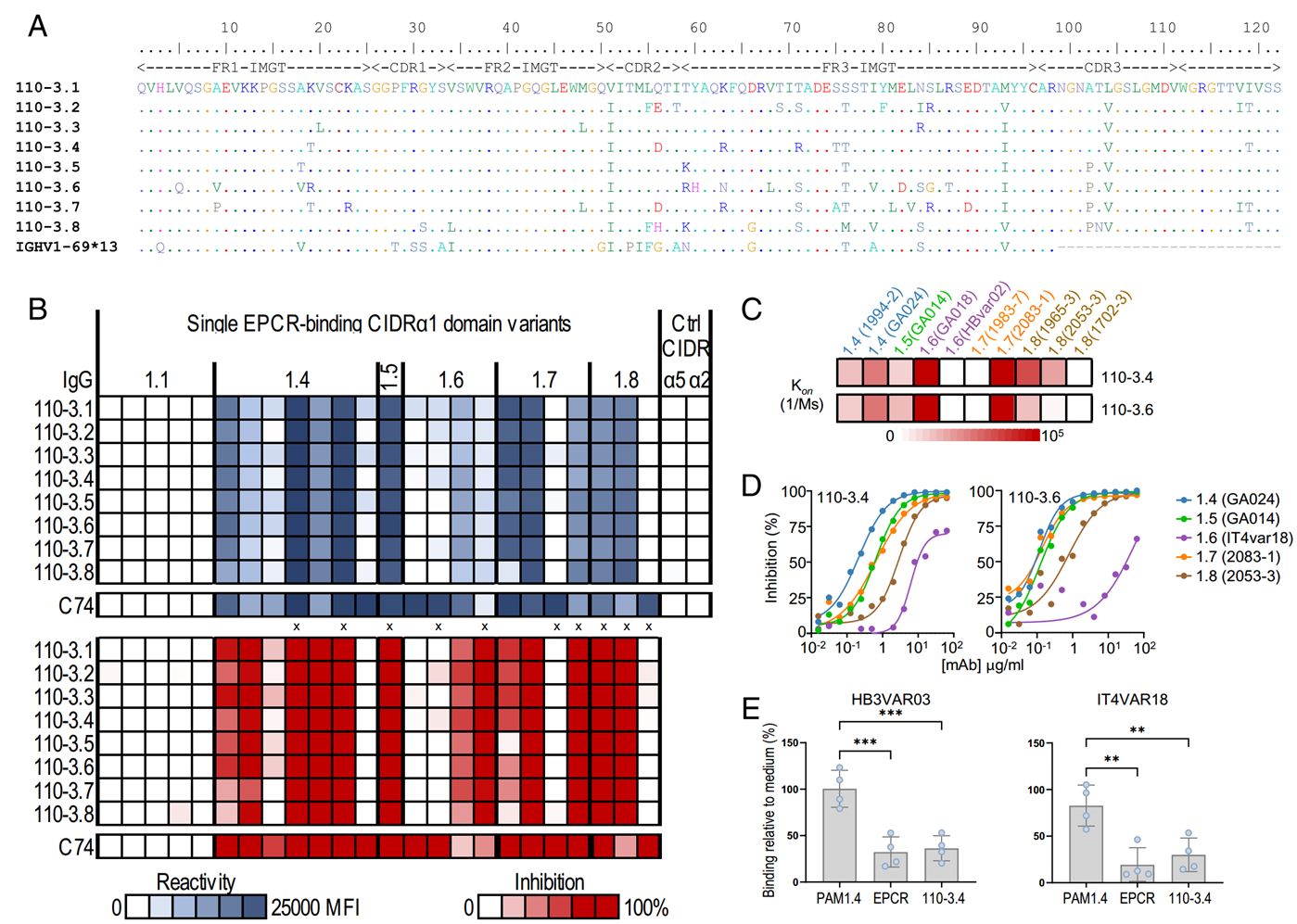
图 2. 110-3家族单克隆抗体的序列和功能分析
中和抗体110-3与CIDRα1结合的结构表征
为揭示作用机制,研究团队通过冷冻电镜解析了110-3.4抗体Fab区与PfEMP1的IT4VAR22三结构域蛋白(含CIDRα1.7)的复合物结构,分辨率达到3.4Å,可以清晰呈现蛋白质的二级结构、抗原抗体互作界面等细节。
结果显示,110-3.4 Fab结合于CIDRα1的EPCR结合及支持螺旋(EB和EBS),其重链CDR3和CDR1形成疏水缝隙,通过氢键与CIDRα1中保守的双苯丙氨酸基序(FF,EPCR结合核心)互补,占据EPCR的结合口袋实现阻断(图 3)。此外,110-3家族的CDR3存在NXT(含糖基化)和NPT(无糖基化)两种基序,但糖基化状态不影响抗体的亲和力和结合广度——冷冻电镜数据显示,110-3.4的聚糖方向远离CIDRα1结合界面,因此不干扰相互作用。
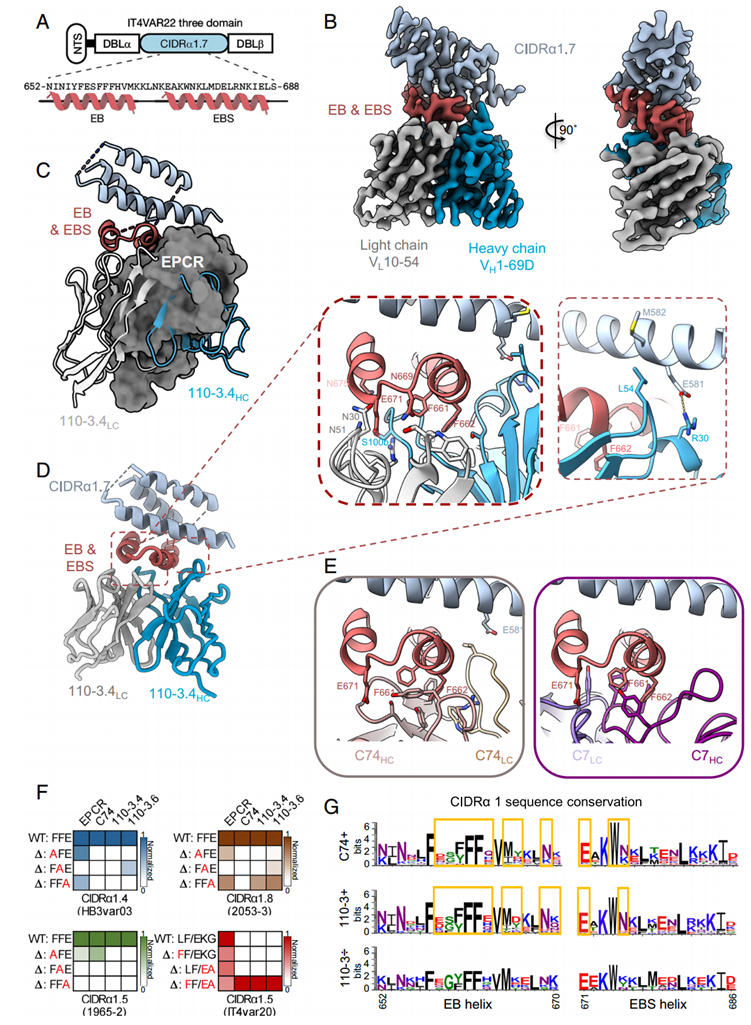
图 3. 110-3与CIDRα1相互作用的分子机制
意义:为疫苗和抗体药物开发提供指导
110-3抗体组是目前第三例报道的CIDRα1广谱抑制性抗体,其发现证实:即便针对CIDRα1这一高度变异的蛋白家族,疟疾暴露人群的免疫系统仍能通过多种分子机制产生靶向同一致病靶点的广谱保护性抗体——110-3使用IGHV1-69基因,与C7、C74的VH基因不同,却都能通过独特的结合模式实现广谱抑制。
这一发现为疟疾疫苗研发提供了新思路:可以借鉴110-3、C7、C74的共同表位(如保守的FF基序),并兼顾它们“和而不同”的作用模式设计疫苗,以诱导更全面的保护屏障。此外,这种从人血清抗体中直接获得的全人源中和抗体,有望直接被开发为抗疟疾药物。更重要的是,这种无需依赖单个B细胞分离筛选、直接从血清中鉴定抗体的技术,为快速挖掘其他传染病(如病毒、细菌感染)的天然保护性抗体开辟了高效路径。或许,解开天然人血清抗体序列的奥秘,正是攻克传染病的关键所在。
参考文献
[1] Turner, L., Nunez de Villavicencio Diaz, T., Raghavan, S. S. R., Kana, I. H., Lyimo, E., Reitzel, C., Wang, C. W., Berube, E., Jensen, R. W., Loeffler, J. R., Fernández-Quintero, M. L., Theander, T. G., Lusingu, J. P. A., Le Bihan, T., Han, X., Minja, D. T. R., Ward, A. B., Ma, B., & Lavstsen, T. (2025). Identification of broadly inhibitory anti-PfEMP1 antibodies by mass spectrometry sequencing of plasma IgG from a malaria-exposed child. Proceedings of the National Academy of Sciences of the United States of America, 122(34), e2508744122. https://doi.org/10.1073/pnas.2508744122
[2] Le Bihan, T., Nunez de Villavicencio Diaz, T., Reitzel, C., Lange, V., Park, M., Beadle, E., Wu, L., Jovic, M., Dubois, R. M., Couzens, A. L., Duan, J., Han, X., Liu, Q., & Ma, B. (2024). De novo protein sequencing of antibodies for identification of neutralizing antibodies in human plasma post SARS-CoV-2 vaccination. Nature communications, 15(1), 8790. https://doi.org/10.1038/s41467-024-53105-8